Le soufre, du fait de sa réactivité intrinsèque, confère aux molécules dans lesquelles il est inséré des propriétés uniques. C'est un élément essentiel pour tous les êtres vivants ; il intervient dans la formule de deux acides aminés naturels, la cystéine et la méthionine et, par conséquent, dans de nombreuses protéines. Cependant, la façon dont le Soufre est inséré dans les biomolécules est une question encore mal comprise en chimie bio-inorganique.
Les mécanismes d’incorporation du soufre dans les biomolécules ne sont que partiellement compris et deux grandes voies réactionnelles sont proposées. La première, bien établie, est la substitution par le sulfure d’un précurseur oxygéné. La deuxième voie, mal comprise, consiste en l’insertion radicalaire directe d’un atome de soufre dans une liaison C-H. À ce jour, on ne connaît que 4 systèmes capables de réaliser ce type d’insertion. Il s’agit de la biotine synthase, de la lipoate synthase et de deux méthylthiotransférases, MiaB et RimO. Ces quatre systèmes sont des protéines à centres FeS capables de réaliser au mieux un seul turnover in vitro. Pour cette raison, il a été proposé et admis que ces systèmes étaient « sacrificiels », c'est-à-dire que le soufre constitutif des centres FeS était celui retrouvé dans le substrat. Ces quatre systèmes appartiennent à la classe des enzymes « Radical SAM » avec comme particularité de contenir deux centres [4Fe-4S], chacun coordonné par 3 cystéines et laissant donc une position de coordination disponible. Le premier centre FeS dit « centre RS » est coordonné par les trois cystéines d’un motif CysXXXCysXXCys caractéristique des enzymes « Radical SAM ». Son quatrième site de coordination est occupé par la S-Adénosylméthionine (SAM). Le deuxième cluster (cluster II) est également lié à trois cystéines et présente lui aussi un quatrième site de coordination disponible.
Afin de comprendre les mécanismes d’insertion du soufre par voie radicalaire, des chercheurs de notre laboratoire se sont intéressés aux méthylthio-transférases MiaB et RimO. Ces enzymes modifient par méthylthiolation une adénine spécifique de certains ARNt ou un résidu aspartate d’une protéine ribosomale (Figure 1).
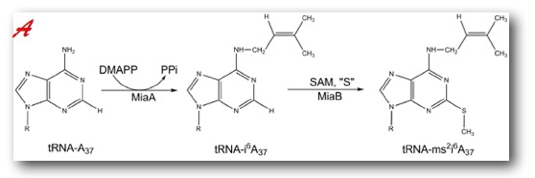
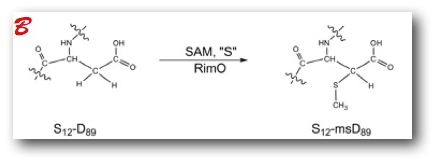
Figure 1. Réactions catalysées par MiaB (A) et RimO (B).
Dans les deux cas, il s’agit d’une réaction chimiquement difficile qui fait intervenir des intermédiaires radicalaires extrêmement réactifs que le système doit contrôler. Pour cette étude, les chercheurs sont partis de l’hypothèse que la fonction du cluster II était de fixer et d’activer un ion sulfure pour générer un radical S• capable de se coupler au substrat lui-même activé sous forme de radical. De fait, les résultats montrent que MiaB et RimO fixent un ion sulfure sur le cluster II (Figure 2, réaction a), puis le méthylent grâce à une première molécule de SAM (Figure 2, réaction b). Enfin, elles transfèrent ce groupement méthylthio au substrat activé grâce à une seconde molécule de SAM fixée au premier cluster RS (Figure 2, réaction c).
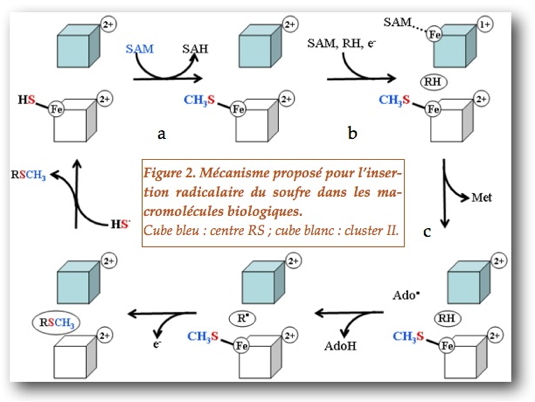
Dans ce scénario le cluster II est capable de fixer et d’activer plusieurs fois de suite un ion sulfure exogène pour, après méthylation, être transféré au substrat permettant d’obtenir un système catalytique et donc de réaliser plus d’un turnover in vitro. Le mécanisme proposé par ces chercheurs est conforté par la structure tridimensionnelle de RimO (Figure 3) qui révèle que les deux centres sont à 8 Å de distance ce qui suggère une forte synergie dans les transferts électroniques impliqués dans ces réactions.
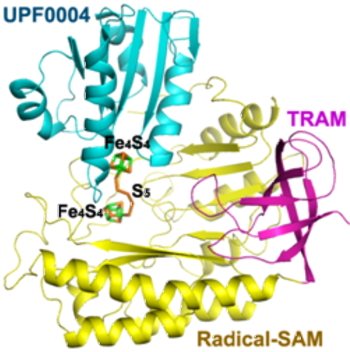
Figure 3. Structure tridimensionnelle de l’enzyme RimO.
En bleu le domaine N-terminal UPF0004 avec le centre Fe-S II, en jaune le domaine Radical-SAM avec le centre Fe-S RS et en C-terminal le domaine TRAM en magenta.
Ces résultats constituent une avancée majeure dans la compréhension des mécanismes d’insertion du soufre dans les macromolécules biologiques.