La lutte contre la résistance des bactéries pathogènes aux antibiotiques constitue l’un des défis sanitaires majeurs. Une nouvelle approche d’anti-virulence consiste à cibler les systèmes biologiques que les bactéries mettent en jeu pour subvenir à leurs besoins en nutriments essentiels. La protéine FUR (Ferric Uptake Regulator) est un régulateur transcriptionnel global qui contrôle l'expression des gènes impliqués dans l'homéostasie du fer, la virulence et le stress oxydatif. Ubiquitaire chez les bactéries Gram-négatives mais absente des eucaryotes, FUR est une cible de premier plan puisque l'inactivation de son gène dans divers pathogènes atténue leur virulence.
Des chercheurs de notre laboratoire, en collaboration avec le Département de Chimie Moléculaire (Université Grenoble Alpes) et l’Institut de Biologie Structurale de Grenoble, mettent au point des inhibiteurs de FUR. Pour réaliser de tels inhibiteurs, ils ont tout d’abord criblé une banque de chimie combinatoire d’aptamères peptidiques pour leur capacité à inhiber la liaison de FUR à l’ADN. Ces aptamères sont constitués d’une boucle peptidique variable de 13 acides aminés constituant la partie active ancrée sur une base protéique constante. Dans un second temps, seule les boucles actives ont a été étudiées sous forme de peptides linéaires de 8 à 13 acides aminés afin de développer des molécules anti-FUR plus petites
[1]. Les chercheurs ont alors identifié les résidus essentiels impliqués dans l’inhibition et dans la zone d’interaction peptide-inhibiteur/protéine cible.
Dans de récents travaux
[2], ils ont étudié les propriétés inhibitrices des peptides les plus actifs précédemment identifiés, afin de caractériser leur interaction avec FUR et de comprendre les mécanismes d’inhibition mis en jeux. Les chercheurs ont été en mesure d’identifier deux types de peptides inhibant soit la dimérisation de FUR soit sa liaison avec l’ADN (
Figure). Par des études alliant mutagenèse, tests d’inhibition, double-hybride, ITC (IsoThermal Calorimetry) et modélisation moléculaire, ils ont identifié les résidus impliqués dans la poche de liaison de FUR à l’ADN et montré que les peptides liés empêchent stériquement l'accessibilité de l'ADN à son site de liaison sur FUR, inhibant efficacement la protéine
[3].
Ces travaux ciblant un système aussi essentiel que la régulation de l’homéostasie du fer chez les bactéries ont permis le développement de petits peptides à action anti-bactérienne. Il sera intéressant de tester la spécificité de ces inhibitions sur les protéines FUR d'autres agents pathogènes ainsi que sur la classe de FUR tétramériques nouvellement identifiées par ces chercheurs
[4].
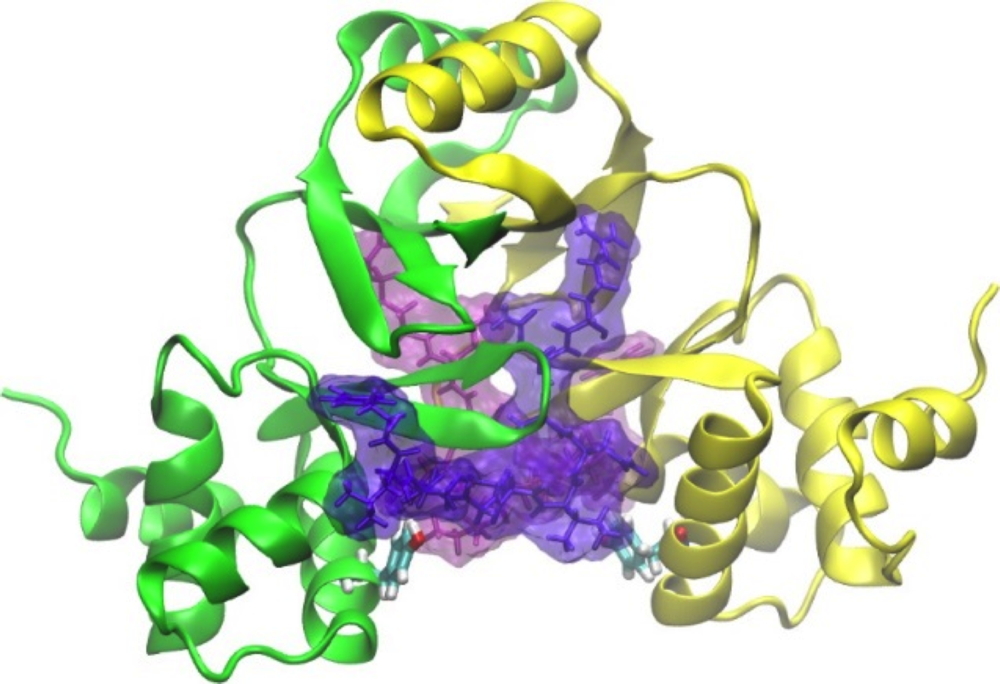
Modèle de structure de 2 peptides inhibiteurs (rose et violet) amarrés simultanément à des sites symétriques sur la protéine FUR d’Escherichia coli et issus du couplage entre expériences d’inhibitions et modélisation. Les chercheurs ont utilisé des expériences de "docking" pour illustrer que ces deux peptides peuvent se lier simultanément à des sites distincts sur le dimère de FUR, un par sous-unité. Jaune : monomère A de FUR ; Vert : monomère B de FUR.
Ces recherches ont été soutenues par une ANR, les LabEx Arcane et GRAL ainsi que par le CEA.