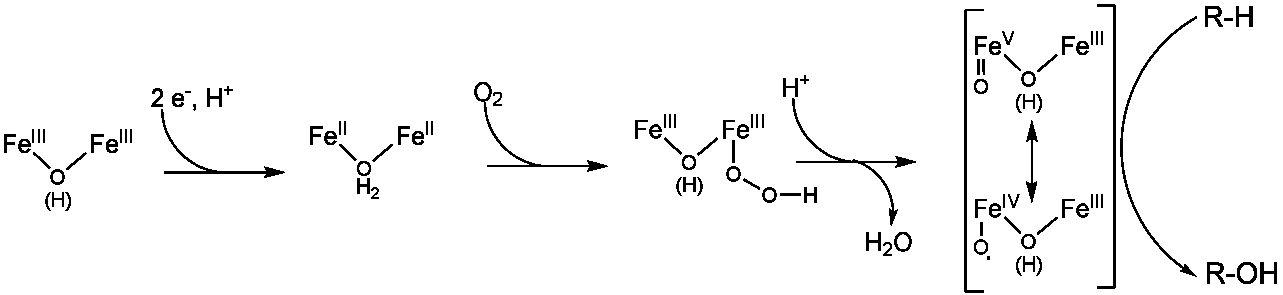
Ces 15 dernières années ont connu une croissance importante du nombre de réactions enzymatiques utilisant la chimie radicalaire dans leurs cycles catalytiques. Les réactions radicalaires en enzymologie sont le plus souvent associées à des réactions dites difficiles en raison de leurs barrières cinétiques et/ou thermodynamiques élevées. Les enzymes impliquées utilisent des cofacteurs rédox qui peuvent être organiques (flavines, ptérines, quinones) ou inorganiques (centres métalliques). Pour fonctionnaliser des liaisons C-H peu réactives ou casser des liaisons C-C, la Nature a mis au point deux stratégies basées sur l’association d’un centre métallique et d’un accepteur d’électrons conduisant à la production d’intermédiaires radicalaires. Ces deux stratégies sont étudiées au laboratoire Chimie et Biologie des Métaux. Dans le premier cas le centre métallique est un complexe de fer (mononucléaire ou binucléaire) et l’accepteur est le dioxygène (Figure 1A). Dans le deuxième cas, le centre métallique est un centre [4Fe-4S] particulier et l’accepteur est une des liaisons C-sulfonium de la S-Adénosylméthionine (SAM) liée au centre Fe-S (Figure 1B). Ce dernier cas sera traité à travers l’exemple de la Spore Photoproduct Lyase (SPL).
A
B
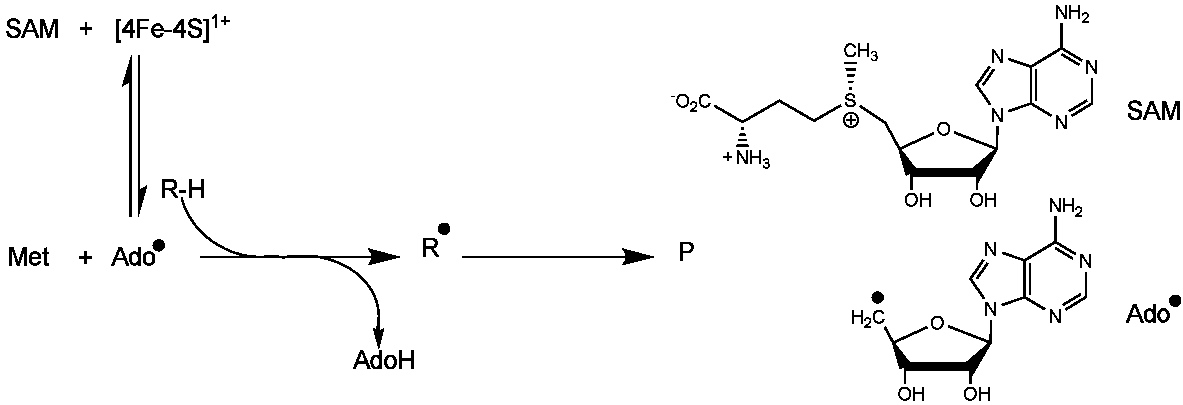
Figure 1 : Mécanisme d’activation de la liaison C-H.
Schéma du mécanisme de fonctionnalisation de la liaison C-H par le système Fe/O2 (A) et Fe-S/SAM (B).
Les centres Fe-S constituent l’un des plus anciens groupes prosthétiques biologiques et assument des fonctions variées. Ils ont été à l’origine reconnus comme médiateurs dans les réactions d’oxydoréduction à un électron. Récemment, une nouvelle fonction pour les centres Fe-S est apparue avec l’identification d’un nombre croissant d’enzymes utilisant un centre [4Fe-4S] en combinaison avec la S-Adénosylméthionine (SAM) pour initier une catalyse radicalaire. De fait, ces enzymes forment une superfamille appelée «Radical-SAM» présente dans les trois domaines du vivant. Toutes les enzymes Radical-SAM ont en commun la production d’un radical 5’-désoxyadénosyle à partir de la SAM servant d’initiateur radicalaire pour la réaction considérée.
Nous nous intéressons à plusieurs enzymes appartenant à cette famille (Figure 2) dont la Spore Photoproduct Lyase, une enzyme de réparation de l’ADN chez les bactéries sporulantes.
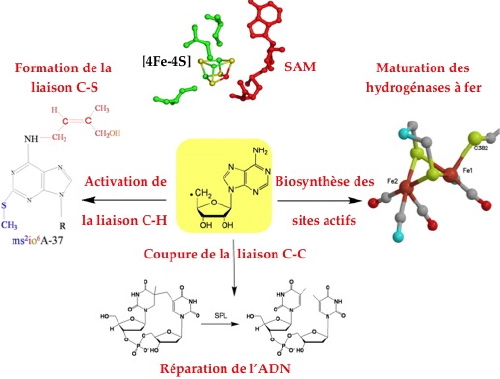
Figure 2 : Production du radical 5-désoxyadénosyle à partir du complexe [4Fe-4S]-SAM pour :
- la transformation de la liaison C-H en liaison C-S.
- la réparation du photoproduit des spores par coupure de la liaison C-C.
- la biosynthèse du site actif de l’hydrogénase à fer.
Les organismes vivants sont en permanence soumis à l'action de rayonnements électromagnétiques et à des agressions chimiques, provenant de molécules toxiques de notre environnement. L’une des cibles biologiques de ces agressions est l’ADN dont les modifications chimiques provoquent des dommages délétères pour la vie cellulaire (mutations, cancer, mort cellulaire). De nombreuses études ont montré que les effets cancérigènes de la lumière solaire étaient principalement dus à l'action du rayonnement UV-B (290-320 nm). À ces longueurs d’onde, les bases de l’ADN absorbent les photons et sont excitées. Il en résulte entre autres des modifications chimiques entre bases pyrimidiques adjacentes conduisant à la formation de photo-produits dimériques nocifs. Pour surmonter ces agressions, les systèmes vivants mettent en œuvre à la fois des systèmes de protection et des mécanismes de réparation. Nombre de ces derniers sont encore mal connus.
En collaboration avec le laboratoire des Lésions des Acides Nucléiques (iNAC/SCIB/LAN, Grenoble) les chercheurs de notre laboratoire essaient de comprendre comment l’association, au sein de la Spore Photoproduct Lyase, d’un centre [4Fe-4S] et de la SAM est mise a profit pour réparer une lésion spécifique de l’ADN. En effet, au moment de la germination, la SPL catalyse la coupure de la liaison C-C au sein d’un dimère de thymine, le photo-produit des spores, résultant de lésions de l’ADN sous l’effet de rayonnement. Ce système illustre comment la Nature utilise des radicaux libres pour réparer une lésion de l’ADN alors que les radicaux sont considérés depuis longtemps comme des espèces toxiques et délétères pour la cellule. Ces chercheurs ont montré qu’il s’agissait en effet d’un contrôle très fin, par l’enzyme, de la production et de l’utilisation d’espèces radicalaires tout au long du cycle catalytique de l’enzyme.
La portée de l’étude menée dépasse largement le cadre des enzymes de réparation de l’ADN et a des prolongements vers la catalyse chimique, la biotechnologie, la microbiologie médicale. En effet, ces recherches ont des retombées importantes dans le domaine thérapeutique avec l’utilisation ciblée de systèmes de réparation sélective dans des situations de stress bien définies ou au contraire à leur inhibition afin de favoriser un processus apoptotique dans la cellule affectée.