L'oxygène est à la fois indispensable et dangereux pour les êtres vivants qui l'utilisent. La réduction de l’oxygène moléculaire en eau par les organismes qui respirent génère des espèces réactives de l'oxygène (ROS), telles que l'anion superoxyde, le radical hydroxyle et le peroxyde d'hydrogène (H2O2). Ces espèces nocives pour les cellules peuvent attaquer les membranes cellulaires, les protéines et les acides nucléiques. En conditions de stress oxydant (concentration anormalement élevée en ROS), les cellules mettent en place des systèmes de détoxication qui reposent essentiellement sur l'expression d'enzymes capables de dégrader les ROS. Par exemple, la superoxyde dismutase (SOD) pour l’anion superoxyde et la catalase pour H2O2.
La protéine PerR (Peroxide resistance Regulator) est un facteur de transcription, senseur spécifique de H2O2, qui régule l’expression des gènes impliqués dans la résistance au stress peroxydique. PerR est une métalloprotéine qui contient deux sites métalliques : un site structural à zinc et un site de régulation ayant une forte affinité pour l’ion Fe2+. En conditions normales, PerR est liée à l’ADN et réprime l’expression des enzymes de défense contre les peroxydes. Lors d’un stress peroxydique, PerR réagit avec H2O2 et le complexe PerR-ADN se dissocie permettant ainsi la levée de répression.
La réaction de PerR avec H2O2 se produit au niveau du site de régulation et conduit à l’oxydation d’un résidu histidine présumé ligand de ce site. Cette oxydation est localisée soit sur l’histidine H37, soit sur l’histidine H91. Il en résulte que la protéine oxydée PerR-Zn-ox est constituée d’un mélange de deux protéines PerR-Zn-H37ox et PerR-Zn-H91ox.
Nous avons comme objectif l’étude biochimique de PerR (Bacillus subtilis) et l’identification de ses changements structuraux depuis la liaison du métal activateur et la fixation de l'ADN jusqu'à la dissociation du complexe ADN-protéine en présence de H2O2. Dans le but de caractériser au niveau structural et fonctionnel la forme oxydée de PerR (PerR-Zn-ox), nous avons été les premiers à exprimer cette protéine de façon exclusive, en grande quantité et sous sa forme physiologique. L’analyse de PerR-Zn-ox en spectrométrie de masse indique que les résidus H37 et H91 sont partiellement oxydés avec une oxydation majoritaire de l’histidine 37 (collaborations M. Jaquinod équipe EDyP du laboratoire Biologie à Grande Échelle de notre institut, et David Lascoux IBS/LSMP). La 2-oxo-histidine a été identifiée comme étant le produit d’oxydation de PerR-Zn-ox. Cette identification a été effectuée par dosage de la 2-oxo-histidine en HPLC-MS/MS (collaboration J-L Ravanat Inac/LCIB). La production en grande quantité de PerR-Zn-ox nous a permis de résoudre sa structure cristallographique, en collaboration avec le Groupe Synchrotron du Laboratoire de Cristallographie et Cristallogenèse des Protéines de l'IBS. Cette structure confirme la présence d’un résidu 2-oxo-histidine en position 37 (Figure 1).
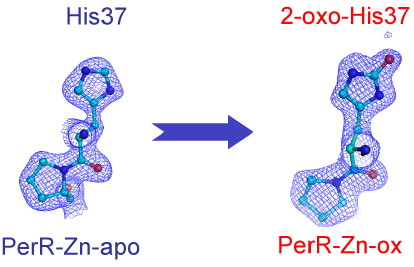
Figure 1 : Représentation du résidu His37 dans la protéine PerR non oxydée (gauche) et du résidu 2-oxo-His37ox dans la protéine PerR-Zn-ox (droite)
La structure de la protéine PerR-Zn-ox, contenant un résidu 2-oxo-histidine, est le premier exemple d’une structure contenant cet acide aminé modifié décrit dans les banques de données de protéines (PDB). La détermination par RPE à température ambiante des constantes d’affinité protéine-métal indique que la forme PerR-Zn-H37ox est capable de lier le métal de régulation alors que la forme PerR-Zn-H91ox ne présente pas d’affinité significative pour ce métal.
De ces résultats, nous proposons deux mécanismes distincts pour l’inactivation de PerR conduisant à la dissociation du complexe protéine-ADN (Figure 2).
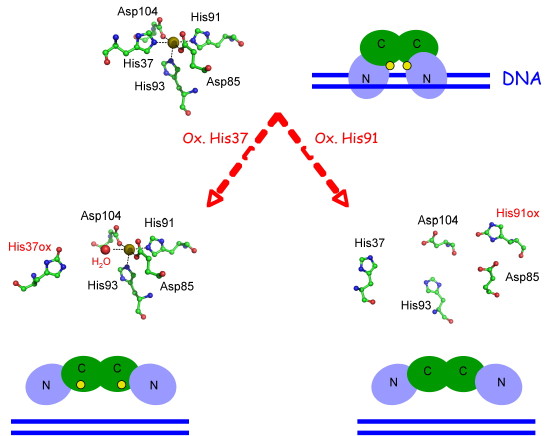
L’ensemble de ces données a permis de comprendre au niveau moléculaire comment la protéine PerR régule la réponse au peroxyde d’hydrogène dans Bacillus subtilis et des bactéries pathogènes.