L’équipe Biocatalyse a été pionnière dans la caractérisation des enzymes à Radical-SAM avec l’étude de l’Activase de la Ribonucléotide Réductase anaérobie d’
E. coli (RNR-AE), un des prototypes de cette classe d’enzymes avec la Lysine Aminomutase (LAM) et l’Activase de la Pyruvate Formate Lyase (PFL-AE)
[Reference]. L’équipe Biocatalyse a étudié par la suite d’autres enzymes à Radical-SAM : la Biotine Synthase, la Lipoate Synthase, la Spore photoproduct Lyase, MiaB, RIMO, …. Les enzymes à Radical-SAM catalysent une grande variété de réactions et actuellement, nos recherches se focalisent sur les enzymes à Radical-SAM impliquées dans des modifications post-transcriptionnelles des ARNt et dans des modifications post-traductionnelles de peptides.
Modification Post-transcriptionnelle des ARNt
M. Atta, V. Duarte, P. Carpentier
Depuis de nombreuses années, l'équipe de BioCatalysis s'intéresse à l'enzymologie de la modification post-transcriptionnelle des ARNt. Les enzymes catalysant la modification des nucléosides sont des cibles attrayantes d’études car elles permettent d’étudier des interactions protéine-ARN. Ces enzymes doivent faire la distinction entre l'ARNt substrat et non-substrat malgré que les ARNt ont tous une forme globalement identique. En outre, les enzymes modifiant l'ARNt catalysent de nombreuses réactions chimiques sophistiquées qui méritent d'être examinées en tant que telles. La voie de biosynthèse favorite de l’équipe concerne la formation du 2-méthylthio-N-6-(cis-hydroxy)isopentényl-adénosine et de ses dérivés (ms
2io
6A
37/ms
2ms
2i
6A
37/ ms
2ms
2io
6A
37) qui est présent en position 37 adjacente à l'anticodon (Figure 1). L'objectif est de comprendre au niveau moléculaire les mécanismes enzymatiques permettant l’insertion des atomes de soufre et d’oxygène dans des liaisons inertes. Ces études mettent en jeu un arsenal de méthodes incluant de la génétique microbienne, de la chimie des protéines et de la spectroscopie avancée.
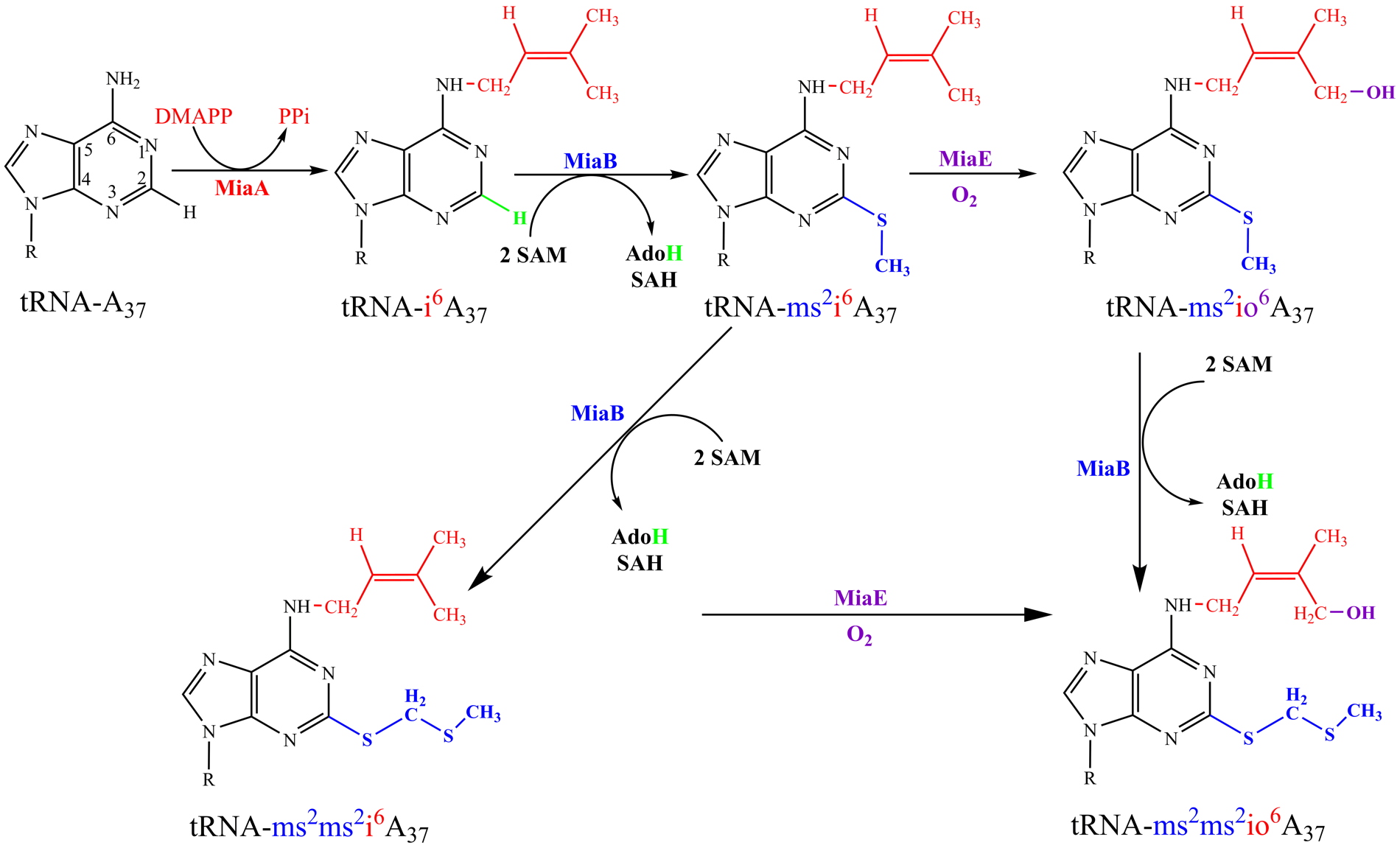
Figure 1 : (A) voie de biosynthèse établie du ms
2io
6A
37. Les enzymes impliquées sont: MiaA, MiaB et MiaE. DMAPP, dimethylallyl diphosphate; PPi, pyrophosphate; SAM, S-adénosylméthionine; SAH, S-adénosylhomocystéine; AdoH, 5’-deoxyadénosyle. (B) La voie de biosynthèse nouvellement découverte du thioacétal ms
2ms
2i
6A
37. (C) Hydroxylation hypothétique du thioacétal par l’enzyme MiaE.
 Selection de publications
Dal Magro C, Keller P, Kotter A, Werner S, Duarte V, Marchand V, Ignarski M, Freiwald A, Muller RU, Dieterich C, Motorin Y, Butter F, Atta M and Helm M
Selection de publications
Dal Magro C, Keller P, Kotter A, Werner S, Duarte V, Marchand V, Ignarski M, Freiwald A, Muller RU, Dieterich C, Motorin Y, Butter F, Atta M and Helm M
A vastly increased chemical variety of RNA modifications containing a thioacetal structure.
Angewandte Chemie-International Edition, 2018,
57(26): 7893-7897
Arragain S, Bimai O, Legrand P, Caillat S, Ravanat JL, Touati N, Binet L, Atta M, Fontecave M, Golinelli-Pimpaneau B and Buckel W
Nonredox thiolation in tRNA occurring
via sulfur activation by a [4Fe-4S] cluster.
Proceedings of the National Academy of Sciences of the USA, 2017,
114(28): 7355-7360
Mulliez E, Duarte V, Arragain S, Fontecave M and Atta M
On the role of additional 4 Fe-4S clusters with a free coordination site in radical-SAM enzymes.
Frontiers in Chemistry, 2017,
5: Article Number 17
Forouhar F, Arragain S, Atta M, Gambarelli S, Mouesca JM, Hussain M, Xiao R, Kieffer-Jaquinod S, Seetharaman J, Acton TB, Montelione GT, Mulliez E, Hunt JF and Fontecave M
Two Fe-S clusters catalyze sulfur insertion by radical-SAM methylthiotransferases.
Nature Chemical Biology, 2013,
9(5): 333-338
Mathevon C, Pierrel F, Oddou JL, Garcia-Serres R, Blondin G, Latour JM, Menage S, Gambarelli S, Fontecave M and Atta M
tRNA-modifying MiaE protein from
Salmonella typhimurium is a nonheme diiron monooxygenase.
Proceedings of the National Academy of Sciences USA, 2007,
104(33): 13295-13300
Modification post-traductionnelle de peptides antimicrobiens : Formation de liaisons thioéther
V. Duarte, M. Atta, C. Basset, L. Shamseddine (PhD CFR CEA 2020-2023)
Il y a quelques années, l'équipe de Biocatalyse a caractérisé l'enzyme Radical-SAM RimO (enzyme du type MiaB) impliquée dans la méthyl-thiolation de la protéine ribosomique S12 d'
E. coli. Actuellement, l'équipe se concentre sur des enzymes Radical-SAM impliquées dans la modification post-traductionnelle de peptides synthétisés par voie ribosomique (RiPPs). Ces enzymes Radical-SAM catalysent un large éventail de modifications telles que des méthylations, la formation de liaisons thioéther et carbone-carbone, des réarrangements complexes et des réactions d’épimérisation. Notre travail concerne la formation de liaisons thioéther entre l’atome de soufre d’une cystéine et le carbone α
¯, ß- ou γ- d'un acide aminé partenaire (Figure 2). Les différences au niveau mécanistique conduisent à deux classes de RiPPs, les Sactipeptides (pont thioéther enC-α) et les Ranthipeptides (ponts thioéther en C-ß ou C-γ).
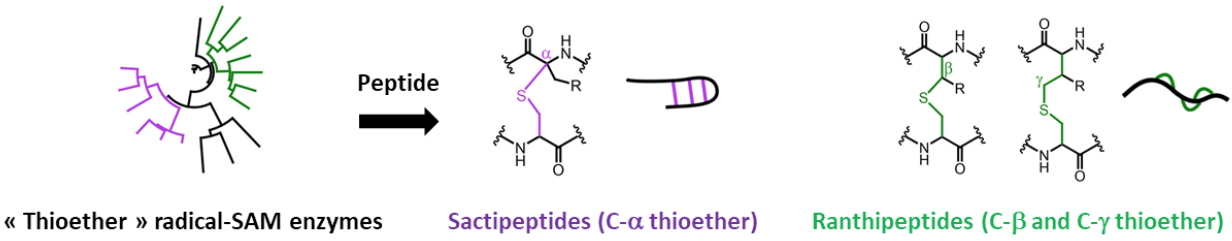
Figure 2 : Les enzymes Radical-SAM dans la biosynthèse des sactipeptides et ranthipeptides.
Les enzymes de la famille Radical-SAM présentent le motif caractéristique CysX
3CysX
2Cys au sein de leur séquence. Ce motif fixe un centre métallique [4Fe-4S]
2+/1+ appelé centre Radical-SAM, dans lequel un atome de Fe libre est utilisé pour lier le cofacteur SAM. À l'état réduit, le centre [4Fe-4S]
1+ catalyse le clivage de la SAM pour générer un radical 5′-deoxyadenosyl (5'-Ado
•). Ce radical, capable d’arracher une atome d’hydrogène du substrat, initie alors différentes réactions chimiques (Figure 3).
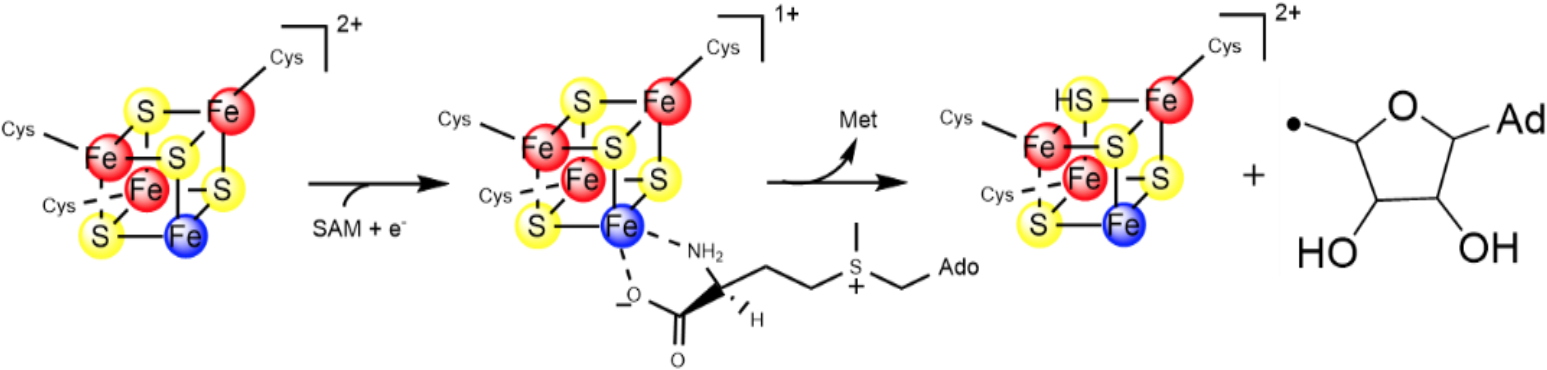
Figure 3 : Coupure de la S-Adénosylméthionine (SAM) par les enzymes Radical-SAM.
En plus du centre [4Fe-4S]
2+ Radical-SAM, la littérature indique que les enzymes impliquées dans la biosynthèse des sactipeptides et des ranthipeptides contiennent dans leur partie C-terminale, un domaine appelé SPASM ou Twitch, contenant un ou plusieurs centres [4Fe-4S]
2+ additionnels. Contrairement aux domaines Twitch, qui ne lient qu'un seul centre Fe-S, les domaines SPASM présentent des séquences riches en Cys capables de coordonner deux centres [4Fe-4S]
2+ supplémentaires. Le rôle de ces centres n’est pas clairement démontré, mais il est suggéré qu'ils interagissent avec le substrat pendant la catalyse ou qu’ils sont impliqués dans des transferts d'électrons. Nos travaux de recherche concernent des nouvelles enzymes Radical-SAM impliquées dans la biosynthèse de peptides antimicrobiens contenant des liaisons thioéther. L'objectif est de comprendre comment ces enzymes installent les modifications post-traductionnelles de manière sélective en : i) déchiffrant le rôle de chaque centre Fe-S; ii) comprenant comment les enzymes interagissent avec le substrat ; iii) étudiant le rôle de la séquence leader présente dans la partie N-terminale des peptides précurseurs. Globalement, ces travaux nous permettront d'explorer la diversité structurale et fonctionnelle des enzymes Radical-SAM et des peptides correspondants.
L'équipe Biocatalyse s’intéresse à la biosynthèse des RiPPs d'un point de vue fondamental mais aussi d'un point de vue santé humaine. En effet, les RiPPs constituent une alternative prometteuse notamment dans la lutte contre la
résistance bactérienne aux antibiotiques.