La découverte de la pénicilline, il y a cent ans, a constitué une avancée médicale majeure qui a lancé le développement des antibiotiques. Cependant, leur utilisation intensive ou inadaptée a conduit à l'émergence de pathogènes résistants ou multi-résistants à toutes les classes d'antibiotiques. Cette situation dramatique conduit les scientifiques d’aujourd’hui à rechercher (i) de nouvelles molécules dont la structure et le mode d'action diffèrent et (ii) de nouvelles cibles afin de prévenir la résistance.
Dans l'équipe Biocatalyse, nous sommes préoccupés par ce problème de la résistance et multi-résistance bactérienne aux antibiotiques (MDR pour « multidrug resistance ») et dans ce contexte, deux projets ont vu le jour au sein de l’équipe. L'un est lié à la
biosynthèse bactérienne du NAD ; l’autre projet concerne les
peptides synthétisés par voie ribosomique et modifiés post-traductionnellement (RiPPs) comme nouvelles molécules contre les souches résistantes et MDR.
Biosynthèse du Nicotinamide Adénine Dinucléotide
S. Ollagnier de Choudens
La voie de biosynthèse du Nicotinamide adénine dinucléotide (NAD) constitue une voie métabolique intéressante pour la découverte et le développement de nouveaux agents antibactériens. Sa biosynthèse implique la formation d'acide quinolinique (QA), dont la biosynthèse diffère chez les procaryotes et les eucaryotes. Chez les eucaryotes, le QA est produit par la dégradation du L-tryptophane, tandis que chez la plupart des procaryotes, le QA est généré à partir du L-aspartate sous l'action concertée de deux enzymes, la L-aspartate oxydase (NadB) et la quinolinate synthase (NadA), une enzyme contenant un centre [4Fe-4S] (Figures 1 et 2). Outre ces synthèses
de novo du NAD, il existe chez certains organismes une voie de secours qui permet le recyclage du NAD (Figure 1). Cependant, certains pathogènes tels que
Mycobacterium leprae et
Helicobacter pylori n'ont pas cette voie de secours. La présence chez les procaryotes et les eucaryotes de voies distinctes pour la biosynthèse du QA, ainsi que l'absence de la voie de secours chez certains pathogènes, font NadA une cible intéressante pour le développement d'agents antibactériens. NadA est une enzyme contenant un centre [4Fe-4S], qui catalyse la condensation entre l'iminoaspartate (IA), généré par NadB à partir du L-asparatate, et la dihydroxyacétone phosphate (DHAP) formant l'acide quinolinique (QA). Ces dernières années, nous avons beaucoup progressé dans la compréhension du mécanisme de la réaction catalysée par NadA en utilisant des approches structurales et biochimiques et nous avons identifié de nouveaux inhibiteurs spécifiques, basés sur le premier inhibiteur découvert (le DTHPA, ACIE en 2012). Actuellement, nos objectifs en ce qui concerne les aspects mécanistiques sont de : (i) piéger les substrats dans les deux cavités du site actif ; (ii) comprendre le mécanisme de condensation, (iii) démêler la stéréochimie de la réaction. En ce qui concerne l'aspect antibactérien, nous visons à (i) optimiser les inhibiteurs connus, dérivés du DTHAP qui ciblent le centre Fe-S ; (ii) concevoir de nouveaux inhibiteurs (qui ne ciblent pas le centre Fe-S) et (iii) étudier la voie de biogenèse du QA chez les bactéries Gram (+), qui utilisent des substrats différents (acétyl-CoA et formiate) et qui fonctionnent probablement selon un mécanisme différent.
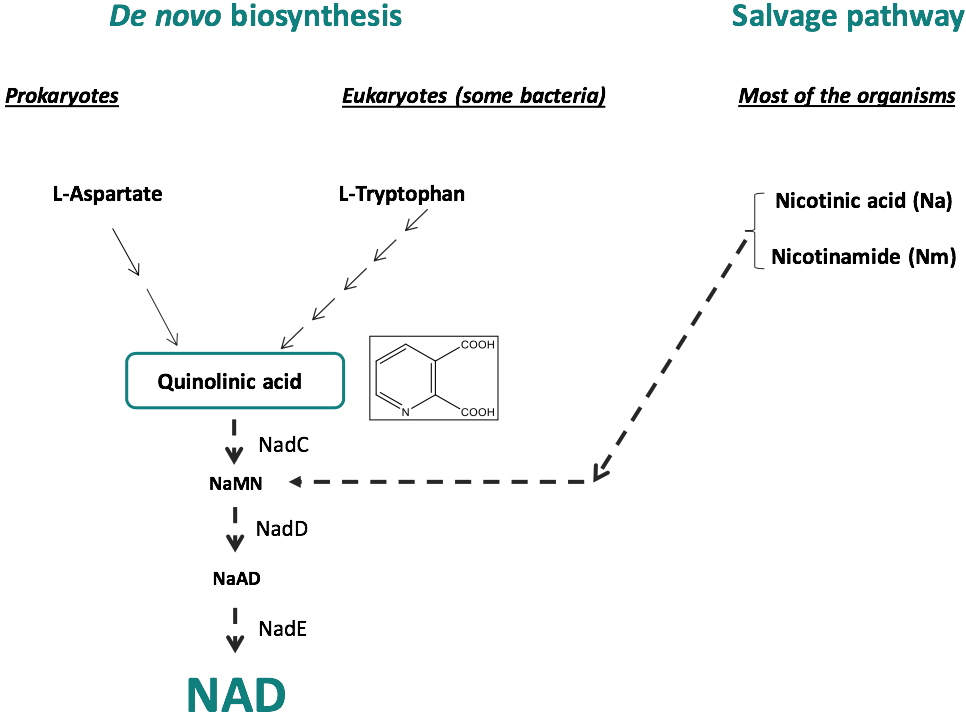
Figure 1 : Biosynthèse du NAD : Biosynthèse
de novo et Voie de secours (Salvage pathway).
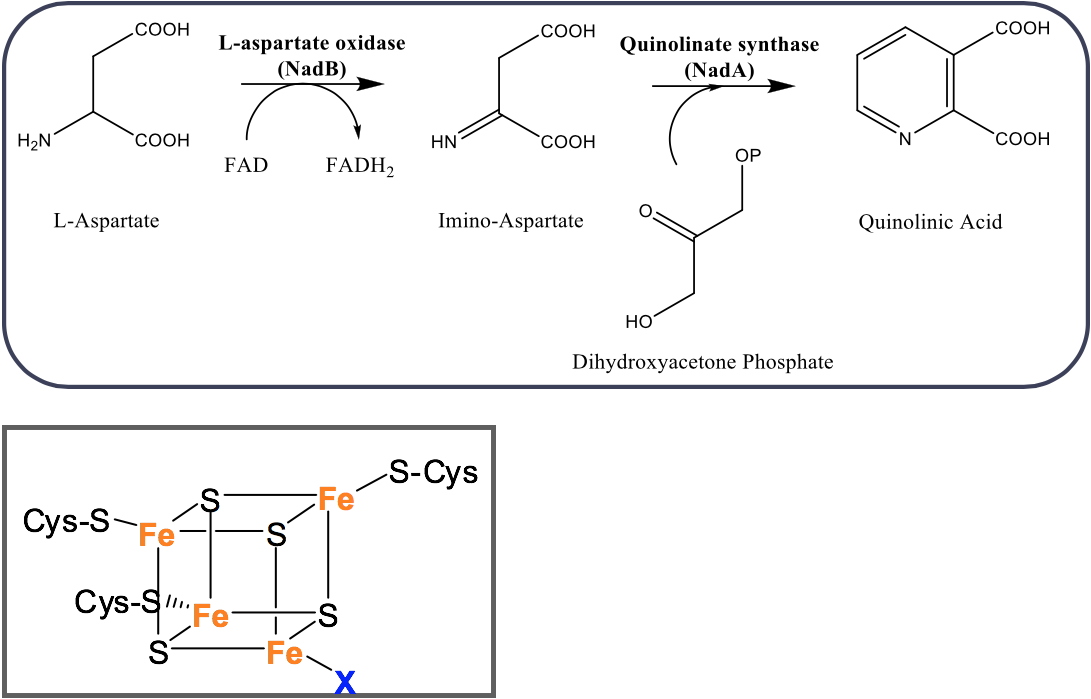
Figure 2 : (A) Formation du QA chez la plupart des procaryotes utilisant NadB et NadA. (B) : Cluster Fe-S cluster de NadA.
 Collaborations
Collaborations
J.C. Fontecilla-Camps (Métalloprotéines, IBS, Grenoble) ; P. Amara (Métalloprotéines, IBS, Grenoble) ; H. de Reuse et S. Cole (Institut Pasteur, Paris) ; O. Hamelin (BioCE, LCBM) ; M.O. Fauvarque (BGE, CEA-Grenoble) ; Y. Sing Wong (DPM, UGA, Grenoble).
 Financements
Financements
Agence nationale de la recherche (ANR NADBiO, ANR NADIN), GRAL Labex, ARCANE Labex.
 Selection de publicationsSaez Cabodevilla J, Volbeda A, Hamelin O, Latour JM, Gigarel O, Clémancey M, Darnault C, Reichmann D, Amara P, Fontecilla-Camps JC and Ollagnier de Choudens S
Selection de publicationsSaez Cabodevilla J, Volbeda A, Hamelin O, Latour JM, Gigarel O, Clémancey M, Darnault C, Reichmann D, Amara P, Fontecilla-Camps JC and Ollagnier de Choudens S
Design of specific inhibitors of quinolinate synthase based on [4Fe-4S] cluster coordination.
Chemical Communications, 2019,
55(26): 3725-3728
Volbeda A, Saez Cabodevilla J, Darnault C, Gigarel O, Han TH, Renoux O, Hamelin O, Ollagnier-de-Choudens S, Amara P and Fontecilla-Camps JC
Crystallographic trapping of reaction intermediates in quinolinic acid synthesis by NadA.
ACS Chemical Biology, 2018,
13(5): 1209-1217
Cherrier MV, Ollagnier de Choudens S and Fontecilla-Camps JC Fe
4S
4 Quinolinate Synthase (NadA).
Encyclopedia of Inorganic and Bioinorganic Chemistry, 2016
Volbeda A, Darnault C, Renoux O, Reichmann D, Amara P, Ollagnier de Choudens S and Fontecilla-Camps JC Crystal structures of quinolinate synthase in complex with a substrate analogue, the condensation intermediate, and substrate-derived product.
Journal of the American Chemical Society, 2016,
138(36): 11802-11809
Reichmann D, Couté Y and Ollagnier De Choudens S Dual activity of quinolinate synthase: Triose phosphate isomerase and dehydration activities play together to form quinolinate.
Biochemistry, 2015,
54(42): 6443-6446
Cherrier MV, Chan A, Darnault C, Reichmann D, Amara P, Ollagnier de Choudens S and Fontecilla-Camps JC The crystal structure of Fe
4S
4 Quinolinate synthase unravels an enzymatic dehydration mechanism that uses Tyrosine and a hydrolase-type triad.
Journal of the American Chemical Society, 2014,
136(14): 5253-5256
Chan A, Clémancey M, Mouesca JM, Amara P, Hamelin O, Latour JM and Ollagnier de Choudens S Studies of inhibitor binding to the [4Fe-4S] cluster of quinolinate synthase.
Angewandte Chemie International Edition, 2012,
51(31): 7711-7714
Rousset C, Fontecave M and Ollagnier de Choudens S The [4Fe-4S] cluster of quinolinate synthase from
Escherichia coli: Investigation of cluster ligands.
FEBS Letters, 2008,
582(19): 2937-2944
Murthy UMN, Ollagnier-de-Choudens S, Sanakis Y, Abdel-Ghany SE, Rousset C, Ye H, Fontecave M, Pilon-Smits EA and Pilon M Characterization of
Arabidopsis thaliana SuFE2 and SuFE3: Functions in chloroplast iron-sulfur cluster assembly and NAD synthesis.
Journal of Biological Chemistry, 2007,
282(25): 18254-18264
Ollagnier-de Choudens S, Loiseau L, Sanakis Y, Barras F and Fontecave M Quinolinate synthetase, an iron-sulfur enzyme in NAD biosynthesis.
FEBS Letters, 2005,
579(17): 3737-3743
Biosynthèse de peptides produits par voie ribosomique et modifiés post-traductionnellement (RiPPs)
V. Duarte, M. Atta, C. Basset, L. Shamseddine (PhD CFR CEA 2020-2023)
La biosynthèse des RiPPs fait intervenir plusieurs familles d'enzymes. Parmi elles, nous nous intéressons aux
enzymes Radical-SAM. Nos travaux concernent les peptides antimicrobiens contenant des liaisons thioéther, à savoir les sactipeptides et les ranthipeptides. Dans la sous-classe des sactipeptides, les enzymes Radical-SAM introduisent des liaisons thioéther intramoléculaires entre le soufre d’une Cys et le carbone α d'un autre acide aminé. Cependant, dans le cas des ranthipepdes, la liaison thioéther implique soit le carbone ß-, soit le carbone γ- du résidu partenaire (Figure 3).
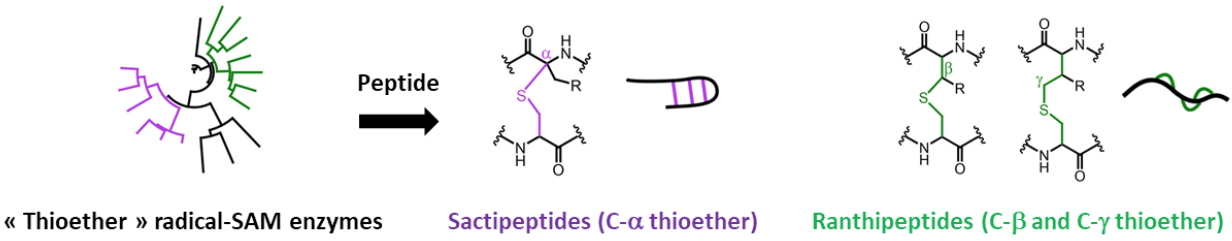
Figure 3 : Les enzymes Radical-SAM dans la biosynthèse des sactipeptides et ranthipeptides.
Nos travaux de recherche concernent des nouvelles enzymes Radical-SAM impliquées dans la biosynthèse de peptides antimicrobiens contenant des liaisons thioéther. Outre l'
intérêt fondamental pour ces enzymes, nous nous intéressons à la biosynthèse des RiPPs d'un point de vue santé humaine. En effet, ces composés constituent une alternative prometteuse notamment dans la lutte contre la résistance bactérienne aux antibiotiques.
De nombreuses études montrent que la microflore intestinale, connue sous le nom de microbiome intestinal, joue un rôle très important dans la santé de l'hôte. Par exemple, la production de bactériocines est l'un des principaux mécanismes de protection mis en œuvre par les bactéries commensales. Nous avons récemment initié en collaboration un projet sur les peptides antimicrobiens RumC produits par
Ruminococus gnavus E1, un symbiote présent dans le tube digestif d’environ 90 % de la population. Notre groupe a développé un système d'expression hétérologue chez
Escherichia coli pour générer un peptide RumC1 strictement identique à celui produit
in vivo par
R. gnavus E1. La production
in vitro de RumC1 était un point crucial. En effet il est nécessaire de produire le peptide en quantité suffisante dans le but de le caractériser au niveau biochimique et structural mais également afin d’évaluer son activité biologique. Nous avons démontré que RumC1 appartient au groupe des sactipeptides. Des expériences de mutagenèse dirigée combinées à des analyses par spectrométrie de masse nous ont permis d'identifier sans ambiguïté les modifications post-traductionnelles de RumC1.
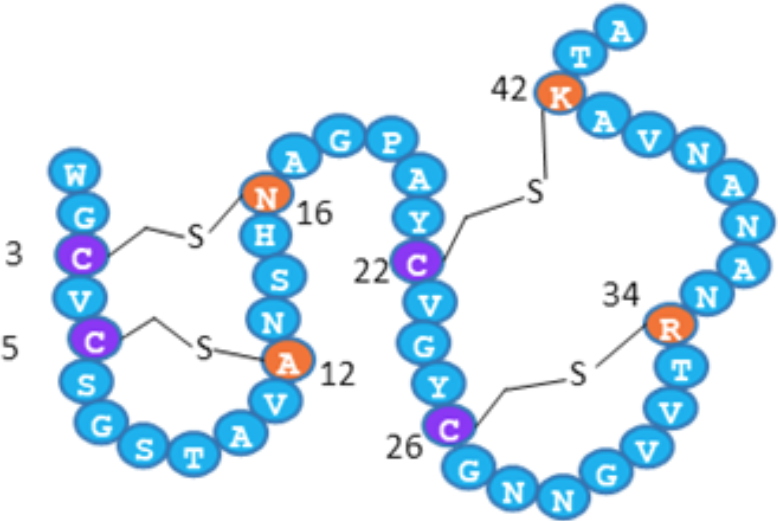
Figure 4 : Modèle structural en double épingle de RumC1. Les résidus Cys, pontés par des liaisons thioéther, sont représentés en violet et leurs partenaires sont indiqués en orange.
La maturation spécifique de RumC1, impliquant l'enzyme Radical-SAM RumMc1, établit un réseau original de quatre liaisons thioéther qui contraint le peptide dans une structure en double épingle (Figure 4). Cette structure confère à RumC1 une activité bactéricide remarquable contre un large éventail de bactéries cibles. En effet, RumC1 est actif à des concentrations inférieures ou égales au micro-molaire sur des souches de laboratoire ou des isolats cliniques de pathogènes Gram-positifs, tels que
Clostridium difficile,
Clostridium perfringens,
Enterococcus faecium et
Streptococcus pneumoniae, y compris des souches multi-résistantes aux antibiotiques courants (Tableau 1).
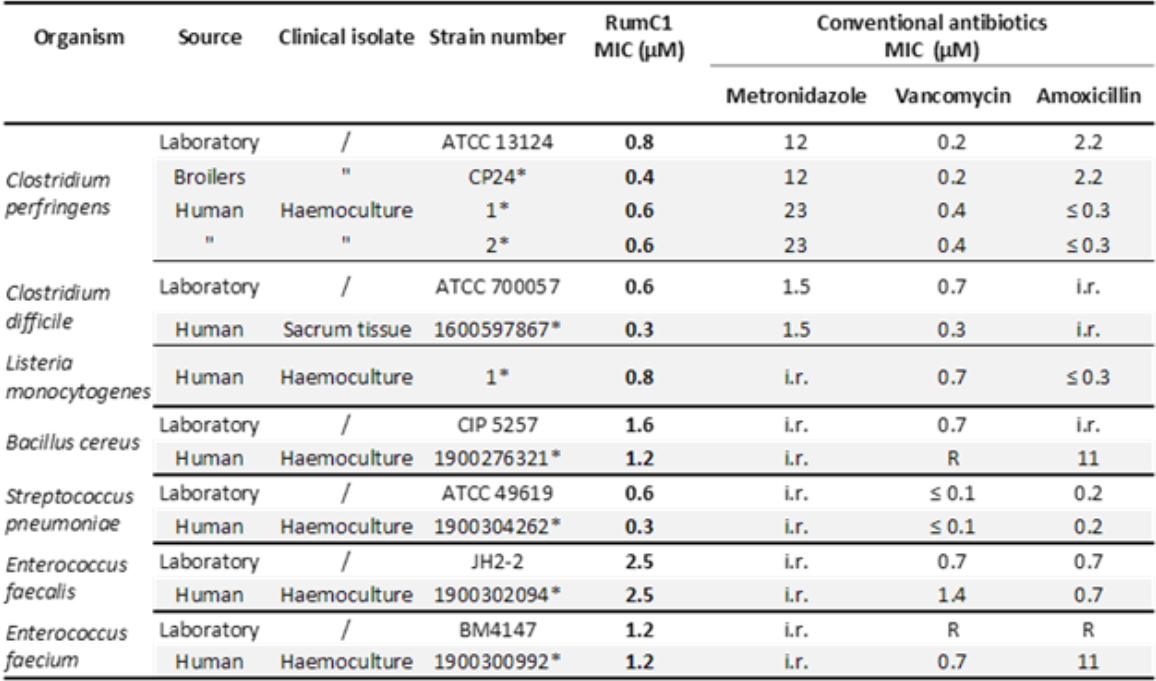
Tableau 1 : Spectre d'activité de RumC1 contre des pathogènes Gram-positifs de laboratoire et des souches cliniques. La résistance acquise aux antibiotiques a été déterminée selon les tableaux de l'EUCAST 2019 et est indiquée par "R", tandis que "i.r." fait référence à la "résistance intrinsèque" et * aux isolats référence de laboratoire.
De façon intéressante, aucun clone bactérien résistant de
C. perfringens, n'est apparu à la suite d'une exposition prolongée à RumC1. En plus de cette efficacité, RumC1 n'est pas significativement toxique sur différentes lignées cellulaires humaines ou explants intestinaux. Enfin, en ce qui concerne le mode d'action de RumC1, ce composé ne crée pas de trous dans la membrane cellulaire et il semble exercer son activité bactéricide sur
C. perfringens en bloquant la synthèse de macromolécules (acides nucléiques, protéines, peptidoglycane).
 Publication Chiumento S, Roblin C, Kieffer-Jaquinod S, Tachon S, Leprètre C, Basset C, Aditiyarini D, Olleik H, Nicoletti C, Bornet O, Iranzo O, Maresca M, Hardré R, Fons M, Giardina T, Devillard E, Guerlesquin F, Couté Y, Atta M, Perrier J, Lafond M and Duarte V
Publication Chiumento S, Roblin C, Kieffer-Jaquinod S, Tachon S, Leprètre C, Basset C, Aditiyarini D, Olleik H, Nicoletti C, Bornet O, Iranzo O, Maresca M, Hardré R, Fons M, Giardina T, Devillard E, Guerlesquin F, Couté Y, Atta M, Perrier J, Lafond M and Duarte V Ruminococcin C, a promising antibiotic produced by a human gut symbiont.
Science Advances, 2019,
5(9): eaaw9969
 Financement
Financement RUMBA project ANR-15-CE21-0020
 Collaborations
Collaborations
M. Lafond, Institut des Sciences Moléculaires de Marseille ; F. Guerlesquin, Institut de Microbiologie de la Méditerranée, Marseille ; E. Devillard, ADISSEO SAS France, Commentry ; S. Torelli, Laboratoire Chimie et Biologie des Métaux, Grenoble ; P. Polard, Lab. de Microbiologie et Génétique Moléculaires, Université Paul Sabatier, Toulouse.